Notch信号通路
Notch信号通路是大多数多细胞生物体中存在的高度保守的细胞信号转导系统。[1]哺乳动物具有四种不同的notch受体,分别称为NOTCH1, NOTCH2, NOTCH3, NOTCH4。Notch受体是单跨膜的受体蛋白。它是由大的胞外部分构成的异源寡聚体,通过钙依赖性的非共价作用与包含有短的胞外结构域,一个跨膜区域和一小段膜内结构域的notch蛋白相互作用。[2]
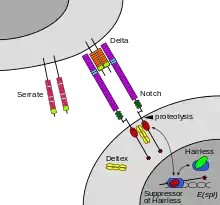
Notch信号通路提升了神经增殖过程中的增殖信号,而其活性被Numb所抑制,以促进神经分化。他在胚胎发育中起到重要的调控作用。
发现
1914年, 约翰·S·德克斯特(John S. Dexter)注意到果蝇(Drosophila melanogaster)的翅膀上有缺口出现。 该基因的等位基因在1917年由托马斯·亨特·摩尔根(Thomas Hunt Morgan)确定。[3][4]其分子层面上的分析和测序由Spyros Artavanis-Tsakonas和迈克尔·扬(Michael Warren Young)在20世纪80年代独立进行。[5][6] 根据发育表型鉴定了两个线虫(C.elegans)中Notch基因的等位基因: lin-12[7] 和 glp-1.[8][9]果蝇lin-12的克隆与部分序列同时由Iva Greenwald报道。[10]
作用机制
Notch蛋白横穿过细胞膜,部分在细胞内而部分在细胞外。配体蛋白结合到胞外域后诱导蛋白切断并释放胞内域,胞内域进而进入细胞膜并调控基因表达。[11]
剪切模型最初在1993年基于在果蝇Notch 和线虫lin-12上的研究而提出。[12][13]对于这一模型,令人信服的证据来自于1998年在果蝇上的活体分析[14]以及细胞培养。.[15] 尽管这个模型在最初引来争议,[1]无可辩驳的证据在2011年被提供。[16][17]
受体通常被直接的细胞-细胞接触所触发。Notch能够使得细胞自我组织-一旦一个细胞表现出一定特性,这一特性可能会通过notch通路被相邻的细胞消除。从而,一群细胞通过相互影响形成一个大结构。由此可见,水平抑制机理对于Notch通路是很关键的。 lin-12 和Notch 介导双细胞的命运决定, 同时水平抑制也包括了放大初始差异的负反馈机制。[16]
Notch 级联包括了Notch和Notch配体以及细胞内把Notch信号转导到细胞核内的蛋白。Notch/Lin-12/Glp-1 受体家族[18] 被发现在果蝇和线虫的细胞命运特化中起作用。[19]
Notch的细胞内结构域与CBF1和Mastermind形成复合物,以激活靶基因的转录。复合体的结构已经确定。
途径
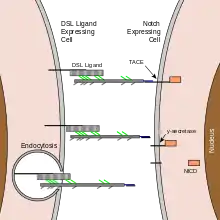
在高尔基体复合体的细胞内运输过程中,切迹受体的成熟涉及预期细胞外侧的裂解。这导致了一种二分蛋白,由一个大的细胞外结构域组成,该结构域与较小的跨膜和细胞内结构域相连。配体的结合促进了两个蛋白水解加工事件;由于蛋白水解,细胞内结构域被释放并可以进入细胞核以与其他 DNA 结合蛋白结合并调节基因表达。
Notch及其大部分配体是跨膜蛋白,因此表达配体的细胞通常必须与表达Notch的细胞相邻才能发生信号传导。Notch 配体也是单通道跨膜蛋白,是 DSL(Delta/Serrate/LAG-2)蛋白家族的成员。在果蝇(果蝇)中,有两种配体,名为 Delta 和 Serrate。在哺乳动物中,相应的名称是类似三角洲和锯齿状的。在哺乳动物中,有多个 Delta 样和锯齿状配体,以及可能的各种其他配体,例如 F3/接触蛋白。
在线虫秀丽隐杆线虫中,两个基因编码同源蛋白 glp-1 和 lin-12。至少有一份报告表明,一些细胞可以发出允许信号传导的过程,这些信号传导发生在相距四到五个细胞直径的细胞之间。
切迹细胞外结构域主要由称为EGF样重复序列的富含胱氨酸的小基序组成。
例如,缺口 1 有 36 个这样的重复。每个 EGF 样重复序列由大约 40 个氨基酸组成,其结构主要由六个保守的半胱氨酸残基定义,这些半胱氨酸残基形成三个保守的二硫键。每个EGF样重复序列都可以被特定位点的O-连接聚糖修饰。[30] 可以在第一和第二保守的半胱氨酸之间添加 O-葡萄糖糖,并且可以在第二和第三保守的半胱氨酸之间添加 O-岩藻糖。这些糖分别由尚未鉴定的 O-葡萄糖基转移酶(Rumi 除外)和 GDP-岩藻糖蛋白 O-岩藻糖基转移酶 1 (POFUT1) 添加。POFUT1 添加 O-岩藻糖对于缺口功能是绝对必要的,并且,如果没有添加 O-岩藻糖的酶,所有缺口蛋白都无法正常发挥作用。到目前为止,缺口糖基化影响功能的方式尚不完全清楚。
通过木糖基转移酶添加两个木糖糖,切口上的O-葡萄糖可以进一步拉长为三糖,而O-岩藻糖可以通过N-乙酰氨基葡萄糖(GlcNAc)糖的有序添加(称为Fringe)的N-乙酰氨基葡萄糖(GlcNAc)糖,通过半乳糖基转移酶添加半乳糖,以及添加唾液酸通过唾液酸转移酶。
为了增加另一个层次的复杂性,在哺乳动物中,有三种边缘 GlcNAc 转移酶,分别称为疯子边缘、躁狂边缘和自由基边缘。这些酶负责对陷波信号传导产生所谓的“边缘效应”。如果 Fringe 在 O-岩藻糖中添加 GlcNAc,则随后会添加半乳糖和唾液酸。在这种四糖存在下,Notch 在与 Delta 配体相互作用时发出强烈的信号,但在与锯齿状配体相互作用时显着抑制信号传导。这种糖的添加抑制通过一种配体的信号传导,并通过另一种配体增强信号传导的方法尚不清楚。
一旦切迹细胞外结构域与配体相互作用,一种称为 ADAM10 的 ADAM 家族金属蛋白酶就会在膜外切割切迹蛋白。这释放了切口的细胞外部分 (NECD),它继续与配体相互作用。然后,配体加上切迹细胞外结构域被配体表达细胞内吞。内吞作用后,配体表达细胞中可能存在信号传导效应;Notch信号转导的这一部分是一个积极研究的主题。[需要引证]在第一次切割之后,一种叫做γ-分泌酶(与阿尔茨海默病有关)的酶切割出表达切迹细胞细胞膜内小叶内的切迹蛋白的剩余部分。这释放了缺口蛋白 (NICD) 的细胞内结构域,然后移动到细胞核,在那里它可以通过激活转录因子 CSL 来调节基因表达。最初认为这些CSL蛋白抑制Notch靶标转录。然而,进一步的研究表明,当细胞内结构域与复合物结合时,它会从抑制因子转变为转录激活因子。其他蛋白质也参与缺口信号级联的细胞内部分。
配体相互作用
当细胞表面的Notch受体与相反细胞上反式呈递的配体结合时,Notch信号转导启动。尽管Notch胞外结构域的大小很大,但已经证明EGF结构域11和12是与Delta相互作用的关键决定因素。其他研究表明 Notch EGF11-12 以外的区域与配体结合有关。例如,Notch EGF 结构域 8 在选择性识别锯齿状/锯齿状中发挥作用,而 EGF 结构域 6-15 是配体刺激时最大信号传导所必需的。Notch1 和 Delta-like 4 (Dll4) 相互作用区域的晶体结构提供了 Notch-配体相互作用的分子水平可视化,并揭示了配体的 N 端 MNNL(或 C2)和 DSL 结构域分别与 Notch EGF 结构域 12 和 11 结合。Notch1-Dll4 结构还阐明了 Notch O 连接岩藻糖和葡萄糖部分在配体识别中的直接作用,并合理化了聚糖介导的 Notch 信号转导调谐的结构机制。
合成Notch信号转导
通过将细胞外受体和细胞内转录结构域替换为其他选择的结构域,可以设计合成Notch受体。这使研究人员能够选择检测哪些配体,以及哪些基因在响应中上调。使用这项技术,细胞可以报告或改变其行为,以响应与用户指定信号的接触,从而促进细胞-细胞信号转导的基础和应用研究的新途径。值得注意的是,该系统允许将多个合成途径并行设计到一个细胞中。
数学建模
Notch-Delta信号转导的数学建模已成为理解细胞间相互作用驱动的模式形成的关键工具,特别是在横向抑制机制的背景下。Collier模型是该领域的基石,它采用耦合常微分方程组来描述相邻细胞之间的反馈回路。该模型由以下公式定义:
和 表示细胞中Notch和Delta活性的水平,函数 和 是典型的希尔函数。反映了信号转导过程中的动态调控。 表示与细胞相邻的细胞的Delta平均活性水平。
该模型的最新扩展包含长程信号传导,承认到达非邻近细胞的细胞突起(如丝状伪足(细胞细胞)的作用。该模型有助于探索模式形成的鲁棒性(稳健性)和生物模式细化,同时考虑丝状伪足动力学和固有噪声的随机性。数学建模在Notch-Delta信号转导中的应用在理解果蝇的切口和翅膀边缘中感觉器官前体(SOP)的模式化方面特别有启发性。
因此,Notch-Delta信号转导的数学建模为生物系统中的横向抑制机制和模式形成提供了重要的见解。它增强了对细胞间相互作用变化的理解,这些变化导致不同的组织结构,有助于发育生物学,并为与Notch-Delta失调相关的疾病提供潜在的治疗途径。
外部链接
- 示意图: notch signaling pathway in Homo sapiens(页面存档备份,存于)
- 示意图:Notch signaling in Drosophila(页面存档备份,存于)
参考文献
- Artavanis-Tsakonas S, Rand MD, Lake RJ. . Science. Apr 1999, 284 (5415): 770–6. Bibcode:1999Sci...284..770A. PMID 10221902. doi:10.1126/science.284.5415.770.
- Brou C, Logeat F, Gupta N, Bessia C, LeBail O, Doedens JR, Cumano A, Roux P, Black RA, Israël A. . Molecular Cell. Feb 2000, 5 (2): 207–16. PMID 10882063. doi:10.1016/S1097-2765(00)80417-7.
- Morgan TH. . The American Naturalist. 1917, 51 (609): 513–544. doi:10.1086/279629.
- Morgan T. revised. Yale University Press. 1928: 77–81 [2017-11-14]. ISBN 0-8240-1384-0. (原始内容存档于2018-06-28).
- Wharton KA, Johansen KM, Xu T, Artavanis-Tsakonas S. . Cell. Dec 1985, 43 (3 Pt 2): 567–81. PMID 3935325. doi:10.1016/0092-8674(85)90229-6.
- Kidd S, Kelley MR, Young MW. . Molecular and Cellular Biology. Sep 1986, 6 (9): 3094–108 [2017-11-14]. PMC 367044
 . PMID 3097517. doi:10.1128/mcb.6.9.3094. (原始内容存档于2019-10-18).
. PMID 3097517. doi:10.1128/mcb.6.9.3094. (原始内容存档于2019-10-18). - Greenwald IS, Sternberg PW, Horvitz HR. . Cell. Sep 1983, 34 (2): 435–44 [2017-11-14]. PMID 6616618. doi:10.1016/0092-8674(83)90377-X. (原始内容存档于2013-10-10).
- Austin J, Kimble J. . Cell. Nov 1987, 51 (4): 589–99 [2017-11-14]. PMID 3677168. doi:10.1016/0092-8674(87)90128-0. (原始内容存档于2018-11-05).
- Priess JR, Schnabel H, Schnabel R. . Cell. Nov 1987, 51 (4): 601–11 [2017-11-14]. PMID 3677169. doi:10.1016/0092-8674(87)90129-2. (原始内容存档于2018-11-05).
- Greenwald I. . Cell. Dec 1985, 43 (3 Pt 2): 583–90 [2017-11-14]. PMID 3000611. doi:10.1016/0092-8674(85)90230-2. (原始内容存档于2018-11-05).
- Oswald F, Täuber B, Dobner T, Bourteele S, Kostezka U, Adler G, Liptay S, Schmid RM. . Molecular and Cellular Biology. Nov 2001, 21 (22): 7761–74. PMC 99946
 . PMID 11604511. doi:10.1128/MCB.21.22.7761-7774.2001.
. PMID 11604511. doi:10.1128/MCB.21.22.7761-7774.2001. - Lieber T, Kidd S, Alcamo E, Corbin V, Young MW. . Genes & Development. Oct 1993, 7 (10): 1949–65 [2017-11-20]. PMID 8406001. doi:10.1101/gad.7.10.1949. (原始内容存档于2018-11-05).
- Struhl G, Fitzgerald K, Greenwald I. . Cell. Jul 1993, 74 (2): 331–45 [2017-11-20]. PMID 8343960. doi:10.1016/0092-8674(93)90424-O. (原始内容存档于2018-11-05).
- Struhl G, Adachi A. . Cell. May 1998, 93 (4): 649–60 [2017-11-20]. PMID 9604939. doi:10.1016/S0092-8674(00)81193-9. (原始内容存档于2018-12-08).
- Schroeter EH, Kisslinger JA, Kopan R. . Nature. May 1998, 393 (6683): 382–6 [2017-11-20]. PMID 9620803. doi:10.1038/30756. (原始内容存档于2019-10-18).
- Greenwald I. . Genetics. Jul 2012, 191 (3): 655–69 [2017-11-20]. PMC 3389966
 . PMID 22785620. doi:10.1534/genetics.112.141812. (原始内容存档于2017-11-10).
. PMID 22785620. doi:10.1534/genetics.112.141812. (原始内容存档于2017-11-10). - Struhl G, Greenwald I. . Proceedings of the National Academy of Sciences of the United States of America. Jan 2001, 98 (1): 229–34. PMC 14573
 . PMID 11134525. doi:10.1073/pnas.98.1.229.
. PMID 11134525. doi:10.1073/pnas.98.1.229. - Artavanis-Tsakonas S, Matsuno K, Fortini ME. . Science. Apr 1995, 268 (5208): 225–32 [2017-11-20]. Bibcode:1995Sci...268..225A. PMID 7716513. doi:10.1126/science.7716513. (原始内容存档于2019-10-18).
- Singson A, Mercer KB, L'Hernault SW. . Cell. Apr 1998, 93 (1): 71–9. PMID 9546393. doi:10.1016/S0092-8674(00)81147-2.