DEAD box
DEAD box為一蛋白質家族的通稱,在原核生物與真核生物細胞中皆有,因其中一個結構域有一天門冬胺酸-麩胺酸-丙胺酸-天門冬胺酸(DEAD)的氨基酸序列而得名[2]。此家族的蛋白多參與RNA相關反應[3],有許多蛋白為RNA解旋酶[4],可以消耗ATP的機制將雙股RNA解開[5]。
| DEAD/DEAH box RNA解旋酶 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 | |||||||||
| 鑑定 | |||||||||
| 標誌 | DEAD | ||||||||
| Pfam | PF00270(旧版) | ||||||||
| Pfam宗系 | CL0023(旧版) | ||||||||
| InterPro | IPR011545 | ||||||||
| PROSITE | PDOC00039 | ||||||||
| SCOP | 1qva / SUPFAM | ||||||||
| CDD | cd00268 | ||||||||
| |||||||||
DEAH家族與SKI2家族的蛋白序列也與DEAD box家族的蛋白相似[6][7][8],三者合稱DExD/H家族蛋白[9]。
結構
1980年代晚期有學者發現許多蛋白的NTP結合位點序列和eIF4ARNA解旋酶的相似[10],後續研究發現這些蛋白均有9個保守的結構域,由N端至C端分別為Q結構域、結構域I、結構域Ia、結構域Ib、結構域II、結構域III、結構域IV、結構域V與結構域VI,其中結構域II又名沃克結構域-B,包含天門冬胺酸-麩胺酸-丙胺酸-天門冬胺酸(DEAD)的氨基酸序列[2]。Q結構域、結構域I、結構域II與結構域VI為結合與水解ATP所需,結構域Ia、結構域Ib、結構域III、結構域IV與結構域V則為結合RNA所需[11]。
功能
DEAD box家族的蛋白多為RNA解旋酶,參與轉錄、RNA剪接、核糖體組裝、RNA轉運、轉譯與RNA降解等細胞中的多種RNA相關反應[9][12][13]。
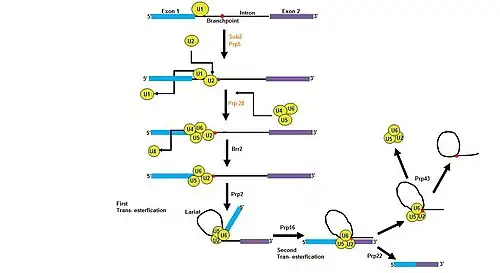
釀酒酵母mRNA剪接的過程有Sub2、Prp28與Prp5等3種DEAD box蛋白參與[5],其中Prp5可與U2 SnRNA結合以影響其結構,使其得以與mRNA內含子中的分支位點(branchpoint)結合[14];Prp28可能可識別內含子的5′端切割位點,但此蛋白無解旋酶活性[15]。另外Prp2、Prp16、Prp22、Prp43與Brr213等多種DEAH家族蛋白也參與mRNA剪切[16]。
轉譯起始因子eIF4A為一DEAD box蛋白,在轉譯起始時可解開mRNA5′ UTR的二級結構,以利核糖體小次單元掃描mRNA以尋找起始密碼子[17]。另一DEAD box Ded1也參與轉譯起始過程,但具體機制仍不明[18];還有一與Ded1相似的DEAD box蛋白Vasa可與eIF2互動[19]。
參考文獻
- Johnson, E. R.; McKay, D. B. . RNA. 1999, 5 (12): 1526–1534. PMC 1369875
 . PMID 10606264. doi:10.1017/S1355838299991410.
. PMID 10606264. doi:10.1017/S1355838299991410. - Linder, P.; Lasko, P. F.; Ashburner, M.; Leroy, P.; Nielsen, P. J.; Nishi, K.; Schnier, J.; Slonimski, P. P. . Nature. 1989, 337 (6203): 121–122. Bibcode:1989Natur.337..121L. PMID 2563148. S2CID 13529955. doi:10.1038/337121a0.
- Takashi Kikuma; Masaya Ohtsu; Takahiko Utsugi; Shoko Koga; Kohji Okuhara; Toshihiko Eki; Fumihiro Fujimori; Yasufumi Murakami. . J. Biol. Chem. March 2004, 279 (20): 20692–20698. PMID 15028736. doi:10.1074/jbc.M400231200
 .
. - Heung LJ, Del Poeta M. . J. Clin. Invest. March 2005, 115 (3): 593–5. PMC 1052016
 . PMID 15765144. doi:10.1172/JCI24508.
. PMID 15765144. doi:10.1172/JCI24508. - Linder P. . Nucleic Acids Res. 2006, 34 (15): 4168–80. PMC 1616962
 . PMID 16936318. doi:10.1093/nar/gkl468.
. PMID 16936318. doi:10.1093/nar/gkl468. - Tanaka N, Schwer B. . Biochemistry. July 2005, 44 (28): 9795–803. PMID 16008364. doi:10.1021/bi050407m.
- Xu J, Wu H, Zhang C, Cao Y, Wang L, Zeng L, Ye X, Wu Q, Dai J, Xie Y, Mao Y. . J. Hum. Genet. 2002, 47 (12): 681–3. PMID 12522690. doi:10.1007/s100380200104
 .
. - Wang L, Lewis MS, Johnson AW. . RNA. August 2005, 11 (8): 1291–302. PMC 1370812
 . PMID 16043509. doi:10.1261/rna.2060405.
. PMID 16043509. doi:10.1261/rna.2060405. - de la Cruz J, Kressler D, Linder P. . Trends Biochem. Sci. May 1999, 24 (5): 192–8. PMID 10322435. doi:10.1016/S0968-0004(99)01376-6.
- Gorbalenya AE, Koonin EV, Donchenko AP, Blinov VM. . Nucleic Acids Res. June 1989, 17 (12): 4713–30. PMC 318027
 . PMID 2546125. doi:10.1093/nar/17.12.4713.
. PMID 2546125. doi:10.1093/nar/17.12.4713. - Tanner NK, Cordin O, Banroques J, Doère M, Linder P. . Mol. Cell. January 2003, 11 (1): 127–38. PMID 12535527. doi:10.1016/S1097-2765(03)00006-6
 .
. - Aubourg S, Kreis M, Lecharny A. . Nucleic Acids Res. January 1999, 27 (2): 628–36. PMC 148225
 . PMID 9862990. doi:10.1093/nar/27.2.628.
. PMID 9862990. doi:10.1093/nar/27.2.628. - Staley JP, Guthrie C. . Cell. February 1998, 92 (3): 315–26. PMID 9476892. S2CID 6208113. doi:10.1016/S0092-8674(00)80925-3
 .
. - Ghetti A, Company M, Abelson J. . RNA. April 1995, 1 (2): 132–45. PMC 1369067
 . PMID 7585243.
. PMID 7585243. - Strauss EJ, Guthrie C. . Nucleic Acids Res. August 1994, 22 (15): 3187–93. PMC 310295
 . PMID 7520570. doi:10.1093/nar/22.15.3187.
. PMID 7520570. doi:10.1093/nar/22.15.3187. - Silverman E, Edwalds-Gilbert G, Lin RJ. . Gene. July 2003, 312: 1–16. PMID 12909336. doi:10.1016/S0378-1119(03)00626-7.
- Sonenberg N. . Prog. Nucleic Acid Res. Mol. Biol. Progress in Nucleic Acid Research and Molecular Biology. 1988, 35: 173–207. ISBN 978-0-12-540035-0. PMID 3065823. doi:10.1016/S0079-6603(08)60614-5.
- Berthelot K, Muldoon M, Rajkowitsch L, Hughes J, McCarthy JE. . Mol. Microbiol. February 2004, 51 (4): 987–1001. PMID 14763975. doi:10.1046/j.1365-2958.2003.03898.x
 .
. - Carrera P, Johnstone O, Nakamura A, Casanova J, Jäckle H, Lasko P. . Mol. Cell. January 2000, 5 (1): 181–7. PMID 10678180. doi:10.1016/S1097-2765(00)80414-1. hdl:11858/00-001M-0000-0012-F80E-6
 .
.