小分子核糖核酸
小分子核糖核酸(英語:,缩写miRNA),又稱微RNA[1](微核糖核酸),是真核生物中廣泛存在的一種長約21到23個核苷酸的RNA分子,可調節其他基因的表达[2][3]。miRNA來自一些從DNA轉錄而來,但無法進一步轉譯成蛋白質的RNA(屬於非編碼RNA)。miRNA通過與目標mRNA结合,進而抑制转录後的基因表达[4],在调控基因表达、细胞周期、生物体发育时序等方面起重要作用。在动物中,一个miRNA通常可以调控数十个基因。
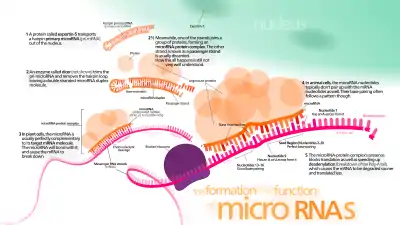
這些RNA是從初級轉錄本(primary transcript)出來的,也就是pri-miRNA,轉變成為稱為pre-miRNA的莖環結構,最後成為具有功能的成熟miRNA。
1989年,Victor发现秀丽隐杆线虫(C. elegans)中有个基因 lin-4 抑制另一个基因 lin-14。他们认为 lin-4 应该也表达一种调控蛋白质,因为基因转录成RNA并翻译成蛋白质是当时认为的公理。不過1993年,Victor的学生 Rosalind Lee 和 Phonda Feinbaum 克隆出了 lin-4,却发现这个基因非常小,不足以做出蛋白質,而且这个基因的产物確實也不是蛋白质,而是一个长度只有22个核苷酸的RNA,後來人們又發現此miRNA可以和其他蛋白質結合,阻斷其他表現,最終產生上述的基因抑制現象。它是由单链的RNA分子产生,这个分子的一端折回来形成不完全的互补配对,称“莖環”[5]。
pri-miRNA长度大约为300~1000个碱基,pri-miRNA经过一次加工后,成为pre-miRNA即microRNA前体,长度大约为70~90个碱基;pre-miRNA再经过Dicer酶酶切后,成为长约20~24nt的成熟miRNA。实际研究中,pre-miRNA应用最早,也最广泛。近年研究发现microRNA的双臂对成熟miRNA的形成有着十分重要的作用。
与小分子siRNAs相比,miRNA在分子特性等方面是相似的,但也存在不少的差异。siRNA是双股RNA,3'端有2个非配对碱基,通常为UU;miRNA是单股RNA。siRNAs是由dsRNA在Dicer酶切割下产生,而成熟miRNAs的产生要复杂一些,首先pri-miRNA在核内由一种称为Drosha酶处理后成为大约70nt的带有茎环结构的Precursor miRNAs(pre-miRNAs),这些pre-miRNAs再在Exportin-5帮助下转运到细胞核外之后再由胞质Dicer酶进行处理,酶切后成为成熟的miRNAs。
生命的一些重要活动如幼虫的生长发育、细胞的发生和分化、神经系统的分化等都被一些非编码蛋白的小RNA的调控,而除miRNA、siRNA以外的小RNA我们目前知之甚少。
命名规则
miR-前缀后面所跟着的数字,代表命名的顺序,比如,miR-124比miR-456发现得早。
“miR-”代表成熟的miRNA、“mir-”代表pre-miRNA和pri-miRNA、“MIR”代表编码miRNA的基因[6]。
miRNA几乎全是独一的编码顺序,但对于拥有一两个碱基不同的则会被标上字母以示,例如,miR-124a与miR-124b。 若成熟的miRNA相同,但pre-miRNA和pri-miRNA和编码他们的基因来自于不同的基因组,则使用数字来表示,例如,mir-194-1和mir-194-2表示两个pre-, pri-miRNA剪切后的成熟miRNA是完全相同的,但却是两个不同的来源。
前缀的三个字母代表了不同的种族来源,例如,hsa-miR-194代表miRNA来源于人类,oar-miR-124来源于绵羊。
对于形成pre-,pri-miRNA莖環的两端miRNA, 通常一端在数量上远远超过另一端。数量优势的一端往往称为guide strand,而另一端被称为passenger strand,通常被大量降解,用*号来表示,例如miR-124和miR-124*。
生物合成機制
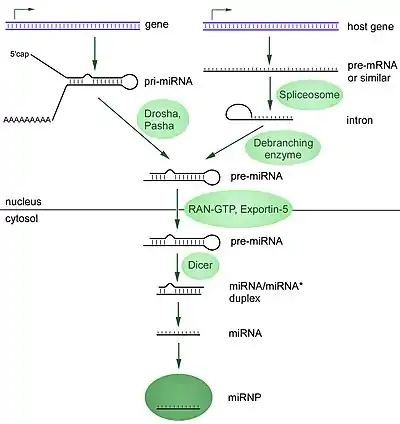
有多達40%的miRNA位於其他基因的內含子或甚至外顯子中[7]。他們通常(但不限於)在有義方向被發現[8][9],因此它們通常與他們宿主基因一起調節[7][10][11]。 位於DNA模板上的序列,並非成熟miRNA的最終編碼:有6%的人類miRNA有RNA編輯的現象,RNA上特定位置的修飾,會產生和他們DNA不同的產物。這增加了miRNA作用的多樣性和範圍,遠超過了基因組單獨引起的作用。
轉錄
miRNA基因通常由RNA聚合酶Ⅱ轉錄[12][13],聚合酶常常會結合到DNA序列附近的啟動子,並將其編碼成前miRNA的髮夾環。 所得到的轉錄產物,上有5'端帽及多聚腺苷酸尾並已被剪接。動物的miRNA最初轉錄為約80個核苷酸的RNA莖環,其會交互形成幾百個核苷酸長的miRNA前體,稱作pri-miRNA[12][8]。當在3'UTR(3'非編碼區)中發現莖-環前體時,該轉錄物可以作為pri-miRNA和mRNA[8]。 此外,RNA聚合酶Ⅲ也會轉錄一些miRNA,特別是具有上游Alu元件的、tRNA或哺乳動物寬分散重複(mammalian-wide interspersed repeats)啟動子單元[14]。
核加工
單個pri-miRNA可以含有1至6個miRNA precursor,這些髮夾環結構各自由約70個核苷酸組成,而每個髮夾的側翼包含了RNA加工的必要序列。 在pri-miRNA中髮夾的雙鏈RNA(dsRNA)結構,會被稱為DiGeorge綜合症關鍵區8(DGCR8或無脊椎動物中的“Pasha”)的核蛋白所識別。隨後DGCR8與Drosha酵素結合形成微加工複合體(Microprocessor complex)[15][16]。
在該複合物中,DGCR8使Drosha的RNase III催化結構區域定向,藉此從髮夾鹼基中切割約11個核苷酸,從而釋放pri-miRNA的髮夾彎[17][18]。所得產物在其3'端具有兩個核苷酸的突出端,其也具有3'羥基和5'磷酸基團。它通常被稱為前miRNA(pre-miRNA)。有許多對於有效加工重要的pre-miRNA下游的序列基序(Sequence motif),已被識別鑑定了[19][20][21]。
而對於那些繞過微加工複合體,直接剪接出內含子的前miRNA,被稱為“Mirtrons”。其最初被認為只存在於果蠅和秀麗隱桿線蟲中,然而現在已經在哺乳動物中發現其存在[22]。 有多達16%的pre-miRNA可以透過核RNA編輯改變[23][24][25],其中最常見地,如腺苷脫氨酶作用於RNA(ADAR)上的腺苷至肌苷(A至I)轉換。另外,RNA編輯也能停止核加工(例如pri-miR-142,其會導致核糖核酸酶Tudor-SN的降解),並改變下游流程包括細胞質miRNA的加工與目標專一性(像是改變中樞神經系統中miR-376的種子區域)[23]。
核輸出
核細胞穿梭蛋白Exportin-5涉及前miRNA髮夾從細胞核輸出的過程。這種蛋白質是karyopherin家族的一個成員,它會識別前miRNA髮夾的3'末端,由RNase III酶與Drosha遺留的兩個核苷酸的突出端。 Exportin-5-mediated介導運輸到細胞質是能量依賴的(主動運輸),其使用GTP來綁定Ran蛋白。[26]
細胞質加工
在細胞質中,前miRNA髮夾會被由RNaseIII酶Dicer所切割[27] ,該內切核糖核酸酶與miRNA髮夾的5'和3'端相互作用並切除連接3'和5'臂的環[28] ,產生長度為22個核苷酸的不完全的miRNA:miRNA*雙鏈體[27] 。整個髮夾長度和環尺寸都會影響Dicer的加工效率。由於RNA的不完全配對性質,miRNA:miRNA*雙鏈體的配對程度也會影響切割[27][29] 。
此外,一些富含G的pre-miRNA可以潛在地利用G-四聯體,來替代典型的莖-環結構。 例如,人的pre-miRNA 92B就使用G-四聯體,來抵抗Dicer在細胞質中的介導切割。[30] 雖然雙鏈體的任一鏈,皆可作為潛在作用的功能miRNA,但通常只有一條鏈會摻入RNA誘導沉默複合體(RISC)中,在其中該miRNA會與其目標mRNA相互作用。
植物中的生物合成
在植物中miRNA的生物合成與動物的最大差異,主要是在於核加工和輸出的過程中:其不像動物使用兩種不同的切割酶(一種位於核內部、一種位於核外部)。植物的兩種切割,都是利用稱為Dicer-like1(DL1)(Dicer同源物)進行,由於DL1僅在植物的細胞核中表達,這表明這兩種切割都在核內發生。在植物miRNA:miRNA *雙鏈體被轉運出細胞核之前,其3'突出端會被稱為Hua-Enhancer1(HEN1)的RNA甲基轉移蛋白甲基化,然後透過稱為Hast(HST)的蛋白質(Exportin 5蛋白的同源物)將雙鏈體從細胞核運出到細胞質中,在那裡它們會分解並且生成成熟的miRNA,來結合到RISC中。[31]
参考文献
- . [2021-07-06]. (原始内容存档于2021-07-09).
- Ambros, V. . Nature. Sep 16, 2004, 431 (7006): 350–5. PMID 15372042. doi:10.1038/nature02871.
- Bartel, DP. . Cell. Jan 23, 2004, 116 (2): 281–97. PMID 14744438. doi:10.1016/S0092-8674(04)00045-5.
- Bartel DP. . Cell. January 2009, 136 (2): 215–33. PMC 3794896
 . PMID 19167326. doi:10.1016/j.cell.2009.01.002.
. PMID 19167326. doi:10.1016/j.cell.2009.01.002. - Ruvkun G(1), Giusto J. . Nature. Mar 1989, 338 (6213): 313–9. PMID 2922060. doi:10.1038/338313a0.
- Wright, MW; Bruford, EA. . Human genomics. Jan 2011, 5 (2): 90–8. PMC 3051107
 . PMID 21296742. doi:10.1186/1479-7364-5-2-90.
. PMID 21296742. doi:10.1186/1479-7364-5-2-90. - Rodriguez A, Griffiths-Jones S, Ashurst JL, Bradley A; Griffiths-Jones; Ashurst; Bradley. . Genome Res. October 2004, 14 (10A): 1902–10. PMC 524413
 . PMID 15364901. doi:10.1101/gr.2722704.
. PMID 15364901. doi:10.1101/gr.2722704. - Cai X, Hagedorn CH, Cullen BR; Hagedorn; Cullen. . RNA. December 2004, 10 (12): 1957–66. PMC 1370684
 . PMID 15525708. doi:10.1261/rna.7135204.
. PMID 15525708. doi:10.1261/rna.7135204. - Weber MJ. . FEBS J. January 2005, 272 (1): 59–73. PMID 15634332. doi:10.1111/j.1432-1033.2004.04389.x.
- Kim YK, Kim VN; Kim. . EMBO J. February 2007, 26 (3): 775–83. PMC 1794378
 . PMID 17255951. doi:10.1038/sj.emboj.7601512.
. PMID 17255951. doi:10.1038/sj.emboj.7601512. - Baskerville S, Bartel DP; Bartel. . RNA. March 2005, 11 (3): 241–7. PMC 1370713
 . PMID 15701730. doi:10.1261/rna.7240905.
. PMID 15701730. doi:10.1261/rna.7240905. - Lee Y, Kim M, Han J, Yeom KH, Lee S, Baek SH, Kim VN; Kim; Han; Yeom; Lee; Baek; Kim. . EMBO J. October 2004, 23 (20): 4051–60. PMC 524334
 . PMID 15372072. doi:10.1038/sj.emboj.7600385.
. PMID 15372072. doi:10.1038/sj.emboj.7600385. - Zhou X, Ruan J, Wang G, Zhang W; Ruan; Wang; Zhang. . PLoS Comput. Biol. March 2007, 3 (3): e37. Bibcode:2007PLSCB...3...37Z. PMC 1817659
 . PMID 17352530. doi:10.1371/journal.pcbi.0030037.
. PMID 17352530. doi:10.1371/journal.pcbi.0030037. - Faller M, Guo F; Guo. . Biochim. Biophys. Acta. November 2008, 1779 (11): 663–7. PMC 2633599
 . PMID 18778799. doi:10.1016/j.bbagrm.2008.08.005.
. PMID 18778799. doi:10.1016/j.bbagrm.2008.08.005. - Lee, Y; Ahn, C; Han, J; Choi, H; Kim, J; Yim, J; Lee, J; Provost, P; Rådmark, O; Kim, S; Kim, VN. . Nature. 25 September 2003, 425 (6956): 415–9. PMID 14508493. doi:10.1038/nature01957.
- Gregory RI, Chendrimada TP, Shiekhattar R; Chendrimada; Shiekhattar. . Methods Mol. Biol. 2006, 342: 33–47. ISBN 1-59745-123-1. PMID 16957365. doi:10.1385/1-59745-123-1:33.
- Han, J; Lee, Y; Yeom, KH; Kim, YK; Jin, H; Kim, VN. . Genes & Development. 15 December 2004, 18 (24): 3016–27. PMC 535913
 . PMID 15574589. doi:10.1101/gad.1262504.
. PMID 15574589. doi:10.1101/gad.1262504. - Han, J; Lee, Y; Yeom, KH; Nam, JW; Heo, I; Rhee, JK; Sohn, SY; Cho, Y; Zhang, BT; Kim, VN. . Cell. 2 June 2006, 125 (5): 887–901. PMID 16751099. doi:10.1016/j.cell.2006.03.043.
- Conrad, Thomas; Annalisa, Marsico; Gehre, Maja; Ørom, Ulf. . Cell Reports. Oct 23, 2014, 9 (2): 542–554. PMID 25310978. doi:10.1016/j.celrep.2014.09.007.
- Auyeung, Vincent; Igor, Ulitsky; McGeary, SE; Bartel, DP. . Cell. Feb 14, 2013, 152 (4): 844–858. PMC 3707628
 . PMID 23415231. doi:10.1016/j.cell.2013.01.031.
. PMID 23415231. doi:10.1016/j.cell.2013.01.031. - Ali PS, Ghoshdastider U, Hoffmann J, Brutschy B, Filipek S. . FEBS Letters. 2012, 586 (22): 3986–90. PMID 23063642. doi:10.1016/j.febslet.2012.09.034.
- Berezikov E, Chung WJ, Willis J, Cuppen E, Lai EC; Chung; Willis; Cuppen; Lai. . Mol. Cell. October 2007, 28 (2): 328–36. PMC 2763384
 . PMID 17964270. doi:10.1016/j.molcel.2007.09.028.
. PMID 17964270. doi:10.1016/j.molcel.2007.09.028. - Kawahara Y, Megraw M, Kreider E, Iizasa H, Valente L, Hatzigeorgiou AG, Nishikura K; Megraw; Kreider; Iizasa; Valente; Hatzigeorgiou; Nishikura. . Nucleic Acids Res. September 2008, 36 (16): 5270–80. PMC 2532740
 . PMID 18684997. doi:10.1093/nar/gkn479.
. PMID 18684997. doi:10.1093/nar/gkn479. - Winter J, Jung S, Keller S, Gregory RI, Diederichs S; Jung; Keller; Gregory; Diederichs. . Nat. Cell Biol. March 2009, 11 (3): 228–34. PMID 19255566. doi:10.1038/ncb0309-228.
- Ohman M. . Biochimie. October 2007, 89 (10): 1171–6. PMID 17628290. doi:10.1016/j.biochi.2007.06.002.
- Murchison EP, Hannon GJ; Hannon. . Curr. Opin. Cell Biol. June 2004, 16 (3): 223–9. PMID 15145345. doi:10.1016/j.ceb.2004.04.003.
- Lund E, Dahlberg JE; Dahlberg. . Cold Spring Harb. Symp. Quant. Biol. 2006, 71: 59–66. PMID 17381281. doi:10.1101/sqb.2006.71.050.
- Park, JE; Heo, I; Tian, Y; Simanshu, DK; Chang, H; Jee, D; Patel, DJ; Kim, VN. . Nature. 13 July 2011, 475 (7355): 201–5. PMID 21753850. doi:10.1038/nature10198.
- Ji X. . Curr. Top. Microbiol. Immunol. Current Topics in Microbiology and Immunology. 2008, 320: 99–116. ISBN 978-3-540-75156-4. PMID 18268841. doi:10.1007/978-3-540-75157-1_5.
- Mirihana Arachchilage G, Dassanayake AC, Basu S. . Chem. Biol. 2015, 22: 262–272. PMID 25641166. doi:10.1016/j.chembiol.2014.12.013.
- Lelandais-Brière C, Sorin C, Declerck M, Benslimane A, Crespi M, Hartmann C; Sorin; Declerck; Benslimane; Crespi; Hartmann. . Current Genomics. March 2010, 11 (1): 14–23. PMC 2851111
 . PMID 20808519. doi:10.2174/138920210790217918.
. PMID 20808519. doi:10.2174/138920210790217918.
外部連結
- miRex: Registry of microRNA expression profiles Internet registry for microRNA expression profiles. The server is part of RNA@IGIB.
- A tutorial on miRNAs by Ambion (页面存档备份,存于)
- 3-D animations describing microRNA formation and function by Rosetta
- Eumir: Web server for prediction of eukaryotic microRNA precursors Web server implementing SVM based prediction of microRNA precursors. Also incorporates companion web server HairpinFetcher.The servers are part of RNA@IGIB.
- 微RNA实验方法 (页面存档备份,存于)